Directeur de recherche CNRS au LaBRI (Laboratoire Bordelais de Recherche en Informatique) et membre du comité de direction VBHI, Pierrick Coupé et son équipe de chercheurs viennent de publier une étude sur une nouvelle méthode basée sur l’imagerie cérébrale et l’intelligence artificielle, appelée arbre de vie du cerveau (lifespan tree of brain anatomy), pour le diagnostic différentiel simultané de plusieurs maladies neurodégénératives. Entretien.
En quoi consiste votre recherche ?
Mon domaine de recherche porte sur la neuroimagerie, notamment sur le développement de nouvelles méthodes d’intelligence artificielle au service des radiologues et neurologues.
Dans cette étude en particulier, nous avons développé un nouvel outil basé sur l’imagerie par résonance magnétique (IRM) et l’intelligence artificielle, appelé arbre de vie du cerveau.
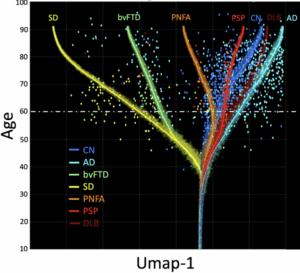
Cet outil modélise l’évolution des structures du cerveau tout au long de la vie (de l’enfance au grand âge) grâce à l’analyse d’un très grand nombre de sujets.
L’idée est de voir comment le cerveau change normalement avec l’âge, et comment il change différemment en cas de maladie neurodégénérative (comme Alzheimer, Parkinson ou d’autres formes de démence).
Une fois que l’on connaît l’évolution moyenne pour chaque pathologie en fonction de l’âge, on peut représenter cette connaissance sous la forme d’un arbre.
Il suffit ensuite de projeter un sujet dans cet espace et de regarder les branches les plus proches pour connaître les maladies potentielles en fonction des modifications observées sur l’IRM.
Pourquoi s’intéresser à l’évolution des structures cérébrales pour diagnostiquer les maladies neurodégénératives ?
Les maladies neurodégénératives présentent souvent des symptômes qui se chevauchent (cognitifs ou moteurs), ce qui complique leur diagnostic clinique.
L’imagerie cérébrale, et notamment l’évolution des structures cérébrales au cours de la vie, contient des informations potentiellement utiles pour distinguer ces maladies.
Cette étude présente une méthode qui modélise ces changements anatomiques (volumes de structures cérébrales obtenus par IRM) sur toute la durée de vie, afin d’aider au diagnostic différentiel entre plusieurs pathologies à la fois.
Concrètement, cette nouvelle méthode modélise les changements de volume de 124 structures cérébrales au cours de la vie afin d’obtenir des représentations interprétables de l’anatomie cérébrale au cours de la progression de la maladie.
Qu’est-ce que le diagnostic différentiel ?
En médecine, le diagnostic différentiel consiste à déterminer quelle maladie précise explique les symptômes d’un patient, surtout quand plusieurs maladies se ressemblent. Par exemple, certains troubles de la mémoire peuvent être liés à Alzheimer, mais aussi à d’autres pathologies.
Le rôle du diagnostic différentiel est d’éviter les confusions et d’orienter vers le bon traitement.
Ici, cela signifie distinguer, par exemple, différentes formes de démence ou différentes causes de parkinsonisme, pour savoir quelle maladie est la plus probable à partir des signatures anatomiques cérébrales.
Pourquoi s’agit-il d’une découverte majeure ?
Aujourd’hui, la très grande majorité des outils disponibles distinguent seulement des patients atteints d’une maladie spécifique de personnes en bonne santé (classification binaire).
Or, pour les cliniciens, la véritable difficulté n’est pas de dire si un patient est malade ou non, mais de savoir quelle maladie exacte il présente.
C’est là que ce nouvel outil apporte quelque chose de réellement utile : il aide à distinguer plusieurs maladies entre elles (classification multi-classes), ce qui est encore très peu exploré en recherche. C’est pourquoi nous avons déposé un brevet en février dernier sur notre approche.
Ensuite, le modèle s’appuie sur l’évolution du cerveau au cours de toute la vie, ce qui donne une représentation plus riche que de regarder une seule IRM à un moment donné.
Ainsi, notre outil n’est pas une “boîte noire” incompréhensible : il fournit des représentations visuelles faciles à lire pour les médecins.
Le problème de l’explicabilité est l’une des principales limites actuelles de l’utilisation de l’IA en médecine. C’est cet aspect qui nous a poussés à choisir une représentation facilement interprétable sous la forme d’un arbre de vie du cerveau.
Quelle est la prochaine étape ?
Tout d’abord, nous allons rendre notre outil accessible gratuitement dans les prochains jours sur la plate-forme volBrain (www.volbrain.net).
Cette plate-forme en libre accès permet de traiter automatiquement des images cérébrales avec tous les outils issus de nos recherches.
Utilisée par plus de 10 000 chercheurs dans le monde, elle a déjà traité plus de 730 000 IRM depuis sa création (volbrain.net/users).
Ensuite, nous travaillons à combiner l’IRM avec d’autres informations médicales (analyses de sang, tests cognitifs, génétique…) pour rendre le diagnostic encore plus précis.
Article scientifique : Coupé, P., B. Mansencal, J. V. Manjón, et al. 2025. Lifespan Tree of Brain Anatomy: Diagnostic Values for Motor and Cognitive Neurodegenerative Diseases. Human Brain Mapping 46, no. 13: e70336. https://doi.org/10.1002/hbm.70336
EM


